MALDI-TOF er et effektivt redskap i diagnostisk fiskebakteriologi
Identifikasjon av bakterier på artsnivå er avgjørende i sykdomsoppklaring for å kunne sette i gang korrekt behandling og smittebegrensende tiltak. Rask avklaring av årsak kan være avgjørende for vellykket håndtering av sykdomsutbrudd. Videre vil diagnostiske metoder basert på bakteriologisk dyrkning være bedre egnet til tidlig oppdagelse av mulige “emerging” bakterielle patogener enn f.eks. PCR-basert diagnostikk.
KUNNSKAP OM FISKEHELSE

I denne spalten vil Veterinærinstituttet i hvert nummer bidra med oppdatert kunnskap om fiskehelse. Ansvarlig for spalten er forsker Mona Gjessing mona.gjessing@vetinst.no
Bakterieisolat kan identifiseres i løpet av få minutter
I diagnostisk arbeid kan langsom vekst hos fiskepatogene bakterier være en kilde til frustrasjon. I tillegg er de også ofte lite reaktive, noe som gjør sikker identifikasjon både tidkrevende og utfordrende med klassisk biokjemisk karakterisering. For en del bakterier er derfor sekvensering av enkeltgener som koder for ribosomale proteiner og/eller andre artsspesifikke gener, nødvendig for en sikker arts-bestemmelse. Ved sekvensering bestemmes koden til genet som undersøkes og sammenlignes med tilsvarende sekvenser i tilgjengelige databaser. Siden tidsfaktoren kan være avgjørende innen diagnostikk, er det også blitt vanlig å bruke qPCR (real time polymerase chain reaction), en metode som identifiserer artsspesifikke gener hos bakterien. Utvikling av sikre spesifikke tester krever at man har god kunnskap både om bakterien man leter etter og de man ikke vil fange opp. Sekvensering og qPCR krever tilgjengelig avansert molekylærbiologiske utstyr og er mer kostbart enn vanlig biokjemisk karakterisering.
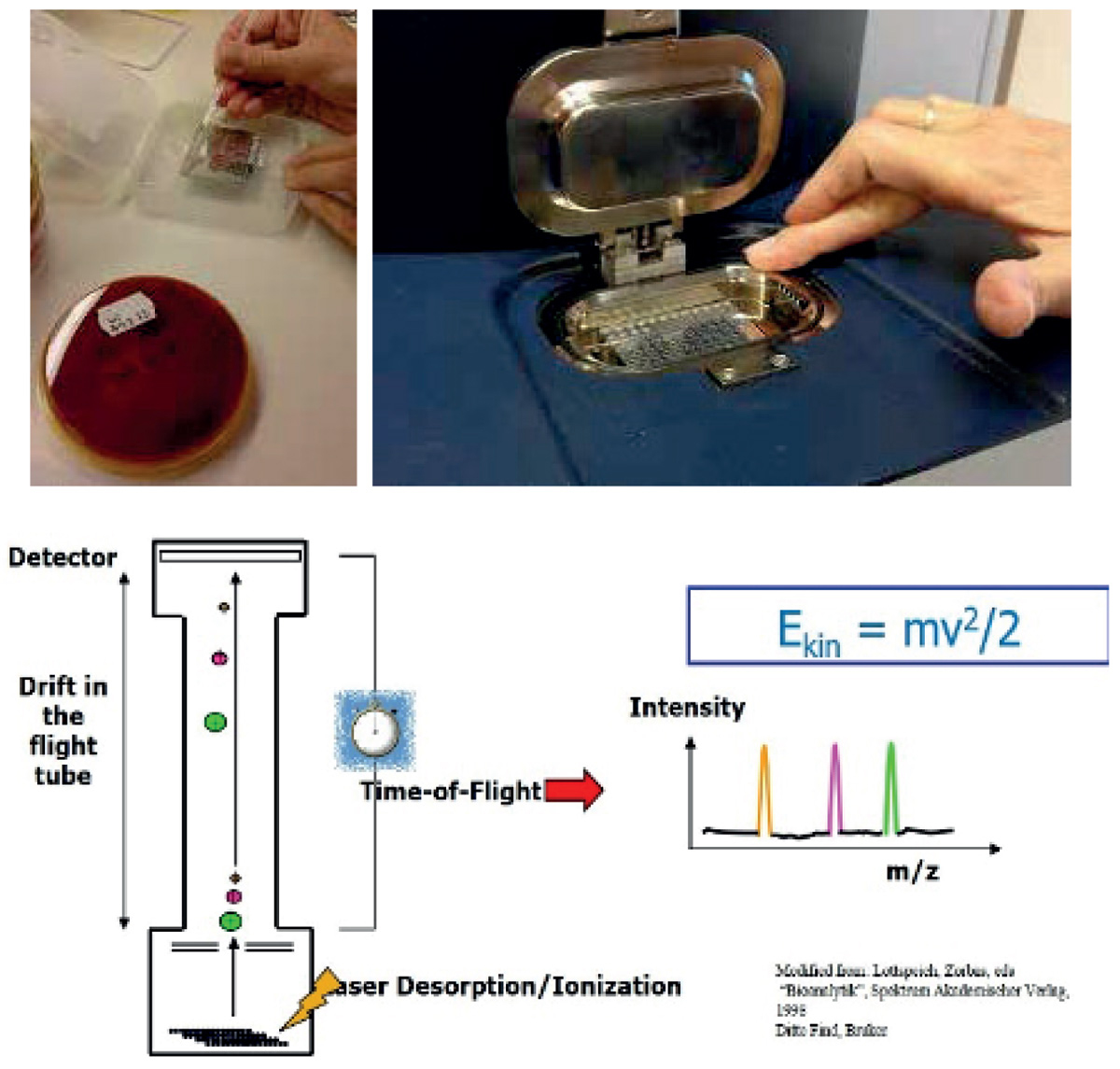
Figur 1. Bakteriemateriale overføres til en stålplate og behandles med matrix som fremmer fordamping og ionisering. Platen plasseres i instrumentets vakuumkammer. Etter tilførsel av energi med laser vandrer bakteriefragmenter gjennom tuben med vakuum til detektorskjermen øverst. Hver gang et ion treffer detektoren lages det et signal. Samlet utgjør signalene et spekter.
I de senere årene er MALDI-TOF MS (Matrix-assisted laser desorption/ ionization time-of-flight mass spectrometry) blitt mer og mer vanlig brukt til rask identifisering av levende bakterier direkte fra bakteriekulturer som vokser på agarskål. «Maldi»-metoden benytter laser til frigjøring av proteiner og massespektrometri til å lage en artspesifikk profil eller ‘fingerprint’ til de enkelte proteinfragmentene i bakterien. Bakteriens ribosomale proteiner har stor betydning for de fragmentmønstrene som genereres.
Bakterien blir identifisert ved å sammenligne profilen med en database hvor det ligger lagret profiler til kjente bakterier. Programvaren graderer eller scorer opp til de 10 mest like profilene i databasen og angir med en score fra 0-3 hvor like de er. En score ≥ 2 mot en kjent bakterie gir god artsidentifikasjon. Metoden er rask å utføre (se figur 1) og identifiserer et bakterieisolat i løpet av få minutter.
VI har etablert profiler for flere fiskepatogener
Naturlig nok er en sikker identifikasjon avhengig av at profilen til bakteriearten som skal bestemmes ligger lagret i databasen. Databasene som finnes på markedet inneholder profiler til en stor mengde bakterier som er viktige i human- og veterinærmedisinen, men fortsatt mangler det kommersielt tilgjengelige profiler til en del fiskepatogene bakterier. Veterinærinstituttet har derfor etablert MALDI-TOF MS profiler for en del vanlig forekommende fiskepatogene bakterier i norsk akvakultur.
Som ved bruk av alle metoder, må resultatene vurderes opp mot eksisterende kunnskap. Hos enkelte bakterieslekter eller genus som f.eks. Vibrio og Aeromonas, vet vi fra arbeid med 16S rRNA sekvenser at de ribosomale proteinene kan være såpass konserverte at koden til disse genene ikke skiller sikkert mellom arter eller underarter innad i genus. Siden det er de ribosomale proteinene som betyr mest for profilene MALDI-TOF MS bruker til artsidentifisering, gjenspeiles dette i resultatene vi får. Førstehånds kunnskap om morfologi, økologisk nisje, og i enkelte tilfeller biokjemiske karakteristika, må derfor alltid vurderes mot resultatene metoden generer.
Gjennom det pågående FHF finansierte prosjektet Tenacibaculum spp. som årsak til atypisk vintersår på norsk oppdrettslaks (Prosjektnr: 901434) har Veterinærinstituttet helgenom-sekvensert et stort antall isolater av Tenacibaculum spp. Bakterier i dette genus er assosiert med sårdannelse hos fisk i norsk akvakultur. Veterinærinstituttet har gjennom et langvarig og grundig arbeid etablert grunnleggende kunnskap om populasjonsstrukturen (slektskap) innad i denne bakterie-slekten. Med dette som bakgrunn utviklet vi profiler til kjente interne og publiserte referansestammer av Tenacibaculum spp. MALDI-TOF grupperingene av et stort antall isolater ble sammenlignet med en helgenomsekvens (WGS-MLST) basert gruppering. Resultatene viste 100 % overensstemmelse mellom MALDI- TOF og WGS sekvensering (figur 2) og validerer dermed MALDI-TOF som en egnet diagnostisk metode, sensitiv nok til å identifisere Tenacibaculum til underarts nivå.
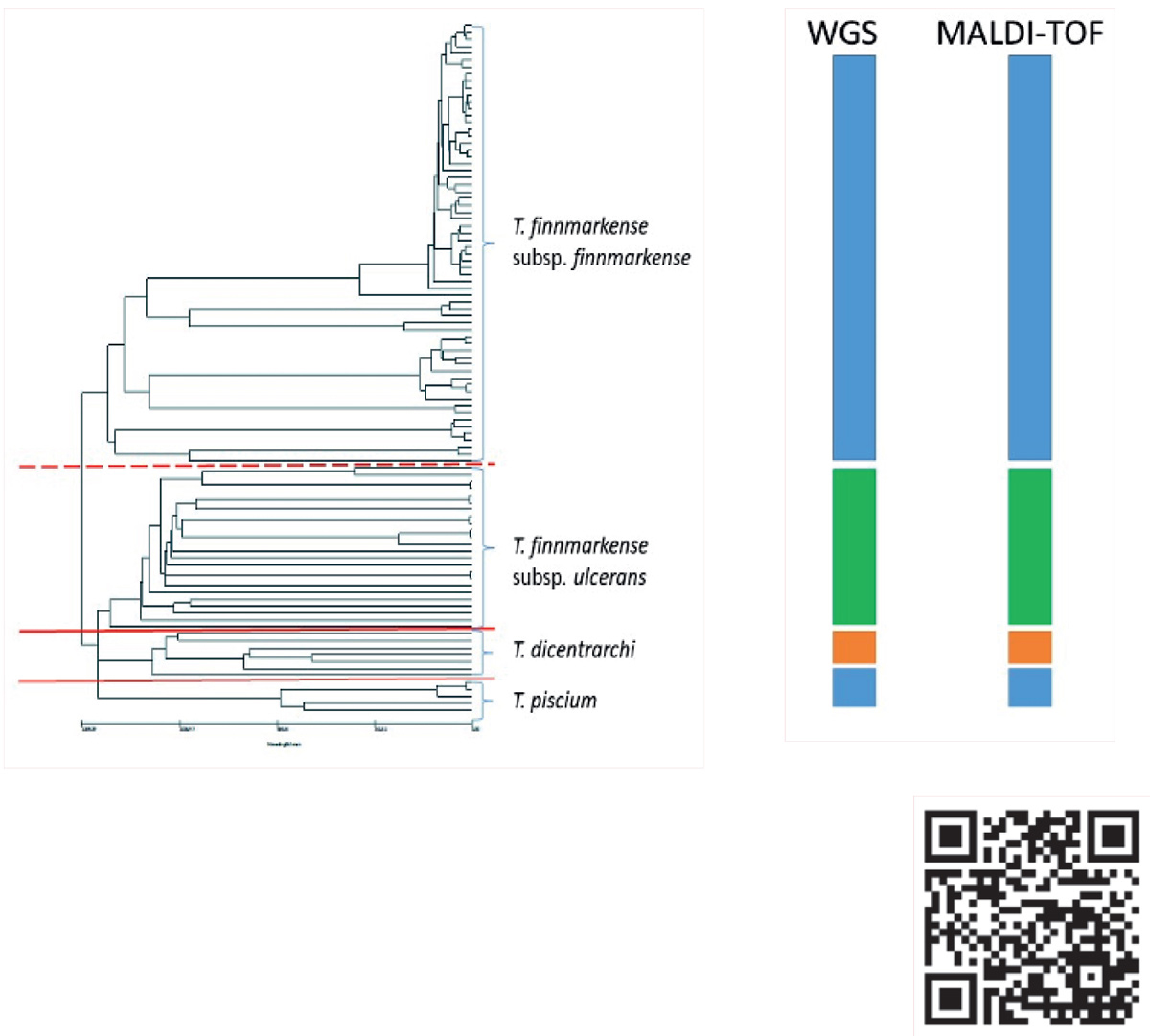
Figur 2. Sammenligning av MALDI-TOF med slektskapsanalyse basert på hel-genom sekvensering. Både artene og sub-artene viser 100% overenstemmelse. Gjennom det pågående FHF finansierte prosjektet Tenacibaculum spp. som årsak til atypisk vintersår på norsk oppdrettslaks (Prosjektnr: 901434) har Veterinærinstituttet helgenom-sekvensert et stort antall isolater av Tenacibaculum spp. Scan QR- koden og les mer.
Veterinærinstituttet har gjennom flere prosjekter, og ved bruk av flere metoder, kartlagt genetisk variasjon hos flere andre fiskepatogene bakterier som bl.a. Pasteurella spp., Moritella viscosa og Yersinia ruckeri. Videre utvikling av MALDI-TOF databasen og sammenligning med resultater fra tidligere genetisk kartlegging vil avklare hvor presise MALDI- TOF profilene er til å bestemme kjente varianter innad i disse bakterieartene. Sannsynligvis vil presisjonen variere på tvers av artene.
Alt i alt er erfaringene med identifikasjon av fiskepatogene bakterier vha. MALDI- TOF gode. Metoden er meget rask og presis og forholdsvis billig i bruk. Tidsbruk for identifikasjon av levende bakteriestammer reduseres fra ca. 2 uker til 0-2 dager og gjør at identifikasjon av bakterieisolater ved dyrkning er tilnærmet like rask som molekylærbiologiske metoder.
MALDI-TOF MS gjør bateriologisk dyrking til en velegnet diagnostisk metode og sikrer at levende bakterier, som senere kan brukes til bl.a. vaksineutvikling og smittesporing, blir isolert fra sykdomsutbrudd. Denne tilnærmingen vil også legge til rette for raskere oppdagelse av mulige ‘emerging’ bakterielle patogener, da dyrkning, i motsetning til PCR, er uspesifikk.